مشروع قاعده بيانات جينوم انسنبل
مشروع قاعدة بيانات الجينوم Ensembl هو مشروع علمى فى المعهد الأوروبى لعلم المعلومات الحيوية ، اللى يوفر مورد مركزى لعلما الوراثة وعلما الأحياء الجزيئية والباحثين التانيين اللى يدرسون الجينومات الخاصة بنوعنا والفقاريات التانيه والكائنات الحية النموذجية .[1][2][3] يعد Ensembl واحد من الكتير من متصفحات الجينوم المعروفة لاسترجاع المعلومات الجينومية . قواعد بيانات ومتصفحات مماثلة فى NCBI وجامعة كاليفورنيا، سانتا كروز (UCSC) .
  | |
|---|---|
| المحتويات | |
| الوصف | Ensembl |
| العناوين | |
| مركز الأبحاث | |
| الوصول | |
| الموقع | www |
| الأدوات | |
| متنوعات | |
تاريخ
تعديلالجينوم البشرى يتكون من 3 مليارات جوز قاعدى ، اللى تشفر يقارب من 20,000 ل25,000 جين . بس الجينوم وحده لا يكون ذا فائدة تذكر، الا اذا نتمكن من تحديد مواقع الجينات الفردية والعلاقات بينها. واحد من الخيارات هو التوضيح اليدوي، حيث يحاول فريق من العلما تحديد الجينات باستخدام البيانات التجريبية من المجلات العلمية وقواعد البيانات العامة. بس، فهذه مهمة بطيئة ومضنية. البديل المعروف باسم التعليق الآلى هو استخدام قوة أجهزة الكمبيوتر للقيام بمدورة الأنماط المعقدة للبروتين مع الحمض النووى .[4][5] تم إطلاق مشروع Ensembl سنة 1999 استجابة للانتهاء الوشيك من مشروع الجينوم البشرى ، و كانت الأهداف الأولية هيا التعليق التلقائى على الجينوم البشري، ودمج ده التعليق مع البيانات البيولوجية المتاحة وجعل كل دى المعرفة متاحة للجمهور.[1] فى مشروع Ensembl، يتم إدخال بيانات التسلسل فى نظام شرح الجينات (مجموعة من "خطوط الأنابيب" البرمجية المكتوبة بلغة Perl ) اللى يقوم بإنشاء مجموعة من مواقع الجينات المتوقعة ويحفظها فى قاعدة بيانات MySQL للتحليل والعرض اللاحق. تجعل Ensembl دى البيانات متاحة مجان لمجتمع الأبحاث العالمي. كل البيانات والرموز اللى أنتجها مشروع Ensembl متاحة للتنزيل، [6] وهناك كمان خادم قاعدة بيانات ممكن الوصول ليه بشكل عام يسمح بالوصول عن بعد. و ذلك، يوفر موقع Ensembl عروض مرئية اتنشأت بواسطة الكمبيوتر لمعظم البيانات.
المشروع مع مرور الوقت، توسع يشمل أنواع إضافية (بما فيها الكائنات النموذجية الرئيسية زى الفأر ، وذبابة الفاكهة ، وسمك الزرد ) و مجموعة أوسع من البيانات الجينومية، بما فيها الاختلافات الجينية والميزات التنظيمية. من ابريل 2009، قام مشروع شقيق، Ensembl Genomes ، بتوسيع نطاق Ensembl ليشمل اللافقاريات ، والنباتات ، والفطريات ، والبكتيريا ، والطلائعيات ، مع التركيز على توفير السياق التصنيفى والتطورى للجينات، فى الوقت نفسه يستمر المشروع الأصلى فى التركيز على الفقاريات.[7][8]
دعمت Ensembl اكتر من 50000 جينوم عبر قواعد بيانات Ensembl وEnsembl Genomes، مضيفة بعض الميزات المبتكرة الجديدة زى Rapid Release ، و هو موقع ويب جديد مصمم لجعل بيانات شرح الجينوم متاحة للمستخدمين بشكل أسرع، و COVID-19 ، و هو موقع ويب جديد للوصول لجينوم مرجعى لـ SARS-CoV-2 .
عرض البيانات الجينومية
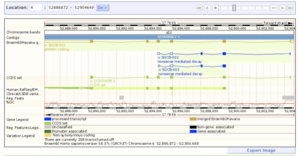
تعديلإن العنصر الأساسى فى مفهوم Ensembl هو القدرة على إنشاء عروض رسومية تلقائى لمحاذاة الجينات والبيانات الجينومية التانيه مقابل جينوم مرجعى . وتظهر دى الصور كمسارات بيانات، ويمكن تشغيل المسارات الفردية و إيقاف تشغيلها،و ده يسمح للمستخدم بتخصيص العرض بما يتناسب مع اهتماماته البحثية. وتتيح الواجهة كمان للمستخدم تكبير منطقة معينة أو التحرك على طول الجينوم فى أى اتجاه.
وتظهر شاشات تانيه البيانات بمستويات متفاوتة من الدقة، بدايه من النمط النووى الكامل وصول لالتمثيلات النصية لتسلسلات الحمض النووى والأحماض الأمينية ، أو تعرض أنواع تانيه من العرض زى أشجار الجينات المتشابهة ( المتماثلات ) عبر مجموعة من الأنواع. ويتم استكمال الرسومات بعروض جدولية، وفى الكتير من الحالات ممكن تصدير البيانات مباشرة من الصفحة فى مجموعة متنوعة من تنسيقات الملفات القياسية زى FASTA .
يمكن كمان إضافة البيانات المنتجة خارجى لالشاشة عن طريق تحميل ملف مناسب ب واحد من التنسيقات المدعومة، زى BAM أو BED أو PSL .
ياتعمل الرسومات باستخدام مجموعة من وحدات Perl المخصصة عن GD ، مكتبة عرض الرسومات القياسية فى Perl.
طرق الوصول البديلة
تعديلبالإضافة لموقعها الإلكتروني، توفر Ensembl واجهة برمجة تطبيقات REST وواجهة برمجة تطبيقات Perl [9] اللى تقوم بإنشاء نماذج للأشياء البيولوجية زى الجينات والبروتينات،و ده يسمح بكتابة نصوص بسيطة لاسترداد البيانات ذات الأهمية. يتم استخدام نفس واجهة برمجة التطبيقات داخلى بواسطة واجهة الويب لعرض البيانات. يتم تقسيمها لأقسام زى واجهة برمجة التطبيقات الأساسية، وواجهة برمجة تطبيقات المقارنة (لبيانات الجينوم المقارنة )، وواجهة برمجة تطبيقات التباين (للوصول لSNPs وSNVs وCNVs ..)، وواجهة برمجة تطبيقات الجينوم الوظيفية (للوصول لالبيانات التنظيمية). يوفر موقع Ensembl معلومات شاملة حول كيفية تثبيت واجهة برمجة التطبيقات واستخدامها . ممكن استخدام البرنامج للوصول لقاعدة بيانات MySQL العامة،و ده يتجنب الحاجة لتنزيل مجموعات بيانات هائلة. ممكن للمستخدمين كمان اختيار استرداد البيانات من MySQL باستخدام استعلامات SQL المباشرة، لكن ده يتطلب معرفة واسعة بمخطط قاعدة البيانات دلوقتى .
يمكن استرجاع مجموعات البيانات الكبيرة باستخدام أداة استخراج البيانات BioMart . إنه يوفر واجهة ويب لتنزيل مجموعات البيانات باستخدام الاستعلامات المعقدة.
وأخير، هناك خادم FTP اللى ممكن استخدامه لتنزيل قواعد بيانات MySQL بالكامل و بعض مجموعات البيانات المحددة بتنسيقات تانيه.
الأنواع دلوقتى
تعديلالجينومات الموضحة تتضمن معظم الفقاريات المتسلسلة بالكامل والكائنات النموذجية المختارة. جميعهم حقيقيات النوى، ومافيش بدائيات النوى. من سنة 2022، هناك 271 نوع مسجل:[10]
شوف كمان
تعديل- قائمة الجينومات حقيقية النواة المتسلسلة
- قائمة قواعد البيانات البيولوجية
- تحليل التسلسل
- أداة تحديد ملف تعريف التسلسل
- نمط التسلسل
- متصفح جينوم جامعة كاليفورنيا سانتا كروز
- ترميز
- 1 فوسفاتيديل اينوزيتول 4
- AADACL3
- AADACL4
- ABAT
- ABCA5
- ABCA7
- ABCA8
- ABCA9
- ABCB4
- ABCC12
- ABCG2
- ABCG5
- ABCG8
- ABI1
- ABI2
- ABI3BP
- ABLIM1
- ABLIM3
- ABTB1
- ACAD10
- ACBD3
- ACIN1
- ACO1
- ACOT13
- ACRV1
- ACSS3
- ACTA2
- ACTG2
- ACTL6B
- ACTL7A
- ACTL8
- ACTR1B
- ACVR2B
- ADAMTS6
- ADAMTSL1
- ADAMTSL4
- ADCK3
- ADD1
- ADD2
- ADD3
- ADM2
- AFAP1
- AFF3
- AFG3L2
- AFTPH
- AGBL4
- AGR3
- AGTRAP
- AGXT2
- AHCTF1
- AHI1
- AHNAK
- AIG1
- AIM1
- AIM2
- AIPL1
- AK3
- AKAP5
- AKAP8L
- AKIP1
- AKIRIN2
- AKR7A3
- AKT1S1
- AKTIP
- ALAS1
- ALG14
- ALPPL2
- ALS2
- ALS2CR8
- ALX1
- ALX4
- ALYREF
- AMOTL2
- AMY1C
- ANAPC2
- ANGPT2
- ANGPT4
- ANGPTL3
- ANKFY1
- ANKH
- ANKRD13C
- ANKRD15
- ANKRD17
- ANKRD25
- ANKRD26
- ANKRD27
- ANKRD31
- ANKZF1
- ANO3
- ANP32B
- ANP32D
- ANP32E
- ANTXR1
- ANTXR2
- AOAH
- AOC2
- AP1AR
- AP1B1
- AP1M1
- AP1M2
- AP1S1
- AP2A2
- AP2B1
- AP2M1
- AP2S1
- AP3B1
- AP3D1
- AP3M1
- AP4B1
- AP4E1
- AP4M1
- APBA1
- APBA2
- APBA2BP
- APBA3
- APBB1IP
- APBB2
- APIP
- APLP1
- APOA1BP
- APOA2
- APOBEC2
- APOBEC3B
- APOBEC3D
- APOBEC3F
- APOBEC3H
- APOBEC4
- APOF
- APOL2
- APOL3
- APOL6
- APOLD1
- APOM
- APPBP2
- APPL1
- APPL2
- AQP6
- ARF1
- ARF3
- ARF4
- ARF5
- ARFGAP3
- ARFGEF1
- ARFGEF2
- ARFIP1
- ARFIP2
- ARFRP1
- ARG2
- ARHGAP11B
- ARHGAP26
- ARHGAP44
- ARHGDIG
- ARHGEF1
- ARHGEF10
- ARHGEF11
- ARHGEF12
- ARHGEF35
- ARHGEF4
- ARHGEF5
- ARHGEF7
- ARID1A
- ARID3A
- ARID3B
- ARID4B
- ARID5B
- ARIH1
- ARL1
- ARL11
- ARL13B
- ARL15
- ARL3
- ARL4A
- ARL6IP1
- ARL6IP5
- ARL8A
- ARL8B
- ARMC5
- ARMC6
- ARMC9
- ARMET
- ARNT2
- ARPC3
- ARPC5
- ARPP 19
- ARV1
- ARVCF
- ASB1
- ASB13
- ASB2
- ASB3
- ASB6
- ASCC2
- ASCC3
- ASF1A
- ASF1B
- ASIC1
- ASIC2
- ASTL
- ASXL1
- ASXL3
- ASZ1
- ATBF1
- ATF5
- ATF6
- ATF7
- ATF7IP
- ATG10
- ATG12
- ATG16L1
- ATG4D
- ATG7
- ATG9A
- ATG9B
- ATOH1
- ATP12A
- ATP1A2
- ATP1A3
- ATP5O
- ATP5SL
- ATP6V0D2
- ATP6V1F
- ATP8B3
- ATP9A
- ATPAF1
- ATRAID
- ATXN2L
- ATXN7L2
- AUTS2
- AVPI1
- AXIN2
- AZGP1
- AZI1
- AZI2
- AZIN1
- Afamin
- Amnionless
- Artemin
- Asporin
- Atlastin
- Attractin
- BABAM1
- BACH1
- BAG2
- BAG4
- BAG5
- BAIAP2L1
- BAIAP3
- BAMBI
- BARHL2
- BASP1
- BAT4
- BAZ1A
- BAZ2A
- BBS10
- BBS12
- BBS2
- BBS4
- BBS7
- BCAP29
- BCAR3
- BCAS1
- BCAS2
- BCAS3
- BCCIP
- BCL11A
- BCL11B
- BCL2L10
- BCL2L11
- BCL2L12
- BCL2L14
- BCL2L2
- BCL6B
- BCL7A
- BCL9
- BCS1L
- BDP1
- BET1
- BET1L
- BFSP1
- BFSP2
- BHLHA9
- BHLHB2
- BICD1
- BICD2
- BIN1
- BIRC6
- BLOC1S1
- BLOC1S2
- BLZF1
- BNIP2
- BNIP3
- BNIP3L
- BNIPL
- BOLL
- BOP1
- BPIFB1
- BPIFB4
- BPTF
- BRAT1
- BRD8
- BRK1
- BRMS1
- BTAF1
- BTBD1
- BTBD14B
- BTBD2
- BTG3
- BTG4
- BTN3A3
- BTNL2
- BYSL
- Bestrophin 2
- Beta actin
- Beta synuclein
- Brevican
- CA1
- CA10
- CAB39
- CAB39L
- CABIN1
- CABLES1
- CABYR
- CACHD1
- CACNA2D3
- CACUL1
- CACYBP
- CADPS
- CADPS2
- CALCOCO1
- CALCOCO2
- CALML3
- CALML5
- CALN1
- CAMK1D
- CAND1
- CAPN1
- CAPN10
- CAPN2
- CAPN5
- CAPN8
- CAPN9
- CARD14
- CASC3
- CASP8AP2
- CASPR
- CASS4
- CASZ1
- CATSPERE
- CBARA1
- CBFA2T2
- CBFA2T3
- CBFB
- CBLC
- CBWD2
- CBX8
- CBY1
- CC2D1A
- CC2D2A
- CCAR1
- CCBE1
- CCDC104
- CCDC137
- CCDC151
- CCDC17
- CCDC18
- CCDC28B
- CCDC3
- CCDC50
- CCDC53
- CCDC55
- CCDC57
- CCDC6
- CCDC70
- CCDC8
- CCDC80
- CCDC85B
- CCDC88A
- CCDC93
- CCM2
- CCNDBP1
- CCNG1
- CCNG2
- CCNL1
- CCP110
- CCT3
- CCT4
- CCT6A
- CCT6B
- CCT7
- CCT8
- CD155
- CD2AP
- CD2BP2
- CD300C
- CD300LF
- CD302
- CD5L
- CDAN1
- CDC123
- CDC16
- CDC23
- CDC34
- CDC40
- CDC42EP1
- CDC42EP2
- CDC42EP3
- CDC42EP4
- CDC73
- CDCA3
- CDCA5
- CDCA7
- CDCA8
- CDH10
- CDH11
- CDH12
- CDH15
- CDH17
- CDH23
- CDH4
- CDH6
- CDH8
- CDH9
- CDK5RAP1
- CDK5RAP3
- CDKAL1
- CDKN2B
- CDKN2C
- CDKN2D
- CDON
- CDX1
- CDYL2
- CEACAM7
- CEBPG
- CECR1
- CEMIP
- CENPC1
- CENPF
- CENPH
- CENPK
- CENPM
- CENPN
- CENPO
- CENPT
- CENTA2
- CENTB2
- CENTD1
- CENTD3
- CENTG1
- CEP104
- CEP120
- CEP128
- CEP135
- CEP152
- CEP164
- CEP192
- CEP250
- CEP350
- CEP55
- CEP57
- CEP63
- CEP68
- CEP70
- CEP72
- CEP76
- CEP78
- CEP97
- CFAP53
- CFC1
- CFDP1
- CFHR1
- CFHR2
- CFHR3
- CFHR4
- CFHR5
- CGB1
- CGB7
- CHAF1A
- CHAF1B
- CHAMP1
- CHCHD2
- CHMP2A
- CHMP2B
- CHMP4B
- CHMP4C
- CHMP5
- CHMP6
- CHODL
- CHRNA6
- CHST14
- CHTF18
- CIAO1
- CIB1
- CIDEA
- CIITA
- CIRBP
- CIRH1A
- CISD2
- CITED2
- CIZ1
- CKAP2
- CKAP5
- CKS2
- CLASP1
- CLASP2
- CLBA1
- CLCA1
- CLCA2
- CLCC1
- CLCF1
- CLCN1
- CLCN3
- CLCN6
- CLCNKB
- CLDN1
- CLDN10
- CLDN11
- CLDN12
- CLDN14
- CLDN17
- CLDN18
- CLDN19
- CLDN20
- CLDN22
- CLDN3
- CLDN4
- CLDN5
- CLDN6
- CLDN7
- CLDN8
- CLDN9
- CLDND1
- CLEC10A
- CLEC11A
- CLEC12A
- CLEC1A
- CLEC1B
- CLEC2B
- CLEC3B
- CLEC4A
- CLEC4C
- CLEC9A
- CLIC5
- CLIC6
- CLIP1
- CLIP2
- CLN5
- CLN6
- CLNS1A
- CLPTM1L
- CLRN1
- CLSPN
- CLSTN1
- CLTB
- CLTC
- CLUH
- CMTM2
- CMTM3
- CNBP
- CNIH
- CNIH4
- CNOT1
- CNOT2
- CNOT3
- CNOT4
- CNOT6
- CNOT7
- CNOT8
- CNTLN
- CNTNAP2
- CNTNAP4
- CNTRL
- CNTROB
- COG1
- COG3
- COG4
- COG5
- COG7
- COMMD1
- COMMD3 BMI1
- COMMD4
- COMMD9
- COPB1
- COPB2
- COPG
- COPS2
- COPS4
- COPS6
- COPS7A
- COPS7B
- COPS8
- COPZ1
- COQ4
- COQ6
- COQ9
- CORO1C
- CORO2A
- COTL1
- COX17
- CPED1
- CPLX2
- CPNE4
- CPNE6
- CPNE8
- CPSF1
- CPSF2
- CPSF3
- CPSF4
- CPSF6
- CPSF7
- CRACR2A
- CRADD
- CRB3
- CREB3
- CREB3L1
- CREB5
- CREBRF
- CREG1
- CRELD1
- CRELD2
- CRHBP
- CRIM1
- CRIP1
- CRIP2
- CRIPT
- CRLF1
- CRLF3
- CRMP1
- CRTC1
- CRTC2
- CRTC3
- CRYAA
- CRYBA4
- CRYBB1
- CRYBB2
- CRYBB3
- CRYGA
- CRYGB
- CRYGC
- CRYM
- CSDC2
- CSMD1
- CSMD2
- CSN2
- CSPG4
- CSPG5
- CSRP1
- CSRP2
- CSRP2BP
- CST1
- CST11
- CST2
- CST4
- CST5
- CST9L
- CSTF1
- CSTF2T
- CSTF3
- CSTL1
- CTA 126B4.3
- CTAGE5
- CTCFL
- CTDNEP1
- CTNNAL1
- CTNNBIP1
- CTNNBL1
- CTNS
- CTTNBP2
- CTTNBP2NL
- CUGBP1
- CUGBP2
- CUL2
- CUL3
- CUL5
- CUL7
- CWC15
- CXXC1
- CXXC5
- CYB561
- CYC1
- CYFIP1
- CYP20A1
- CYP26A1
- CYP26B1
- CYP26C1
- CYP27C1
- CYP2A13
- CYP2A7
- CYP2C18
- CYP2W1
- CYP39A1
- CYP3A43
- CYP4A22
- CYP4B1
- CYP4F22
- CYP4V2
- CYP4X1
- CYP4Z1
- CYP8B1
- CYTH2
- CYTH3
- CYTH4
- CYTL1
- CYYR1
- Calcyphosin
- Calmegin
- Calpastatin
- Calumenin
- Caprin 1
- Cochlin
- Cofilin 2
- Coilin
- Consortin
- Corneodesmosin
- DAAM1
- DAB2IP
- DAPP1
- DAZAP2
- DAZL
- DBC1
- DBNDD2
- DBR1
- DBX1
- DBX2
- DCBLD2
- DCC1
- DCDC2
- DCHS1
- DCHS2
- DCLRE1A
- DCLRE1B
- DCP1A
- DCP1B
- DCP2
- DCTN1
- DCTN3
- DCTN4
- DCTN5
- DCUN1D1
- DCUN1D4
- DDEF1
- DDEF2
- DDI1
- DDX19B
- DDX27
- DDX56
- DEC1
- DEDD
- DEF6
- DEFA1
- DEFA1B
- DEFA3
- DEFA6
- DEFB103A
- DEFB118
- DEFB119
- DEFB126
- DEFB127
- DEFB129
- DEGS2
- DENND1A
- DENND4A
- DEPDC1
- DEPTOR
- DFNA5
- DGCR14
- DGCR2
- DGCR6
- DGCR8
- DHRS7
- DHTKD1
- DHX29
- DIAPH1
- DIDO1
- DIP2A
- DIRC2
- DIS3L2
- DIXDC1
- DKK2
- DKK3
- DLG5
- DLGAP1
- DLGAP2
- DLGAP4
- DLGAP5
- DLK1
- DLL3
- DLL4
- DMBT1
- DMP1
- DMRT1
- DMTF1
- DMXL2
- DNAAF2
- DNAH1
- DNAH5
- DNAH7
- DNAH9
- DNAI1
- DNAI2
- DNAJA1
- DNAJA2
- DNAJA4
- DNAJB1
- DNAJB11
- DNAJB2
- DNAJB4
- DNAJB9
- DNAJC1
- DNAJC10
- DNAJC11
- DNAJC14
- DNAJC28
- DNAJC7
- DNAL1
- DNAL4
- DNER
- DNM3
- DOC2A
- DOK2
- DOK3
- DOK4
- DOK5
- DOM3Z
- DOT1L
- DPAGT1
- DPF1
- DPF2
- DPM2
- DPP10
- DPP6
- DPY19L2
- DRAP1
- DRD1IP
- DRG1
- DRG2
- DSC1
- DSN1
- DTNA
- DTNB
- DTX1
- DUSP6
- DUX4
- DVL2
- DVL3
- DYM
- DYNC1H1
- DYNC1I1
- DYNC1I2
- DYNC1LI1
- DYNC1LI2
- DYNC2H1
- DYNLL2
- DYNLRB1
- DYNLT1
- DZIP1
- Derlin 1
- Derlin 2
- Derlin 3
- Dermatopontin
- Desmoglein 1
- Desmoglein 3
- Desmoglein 4
- Dock10
- Dock5
- Dock6
- Dock9
- Drebrin like
- EAF1
- EAF2
- EAPP
- EBF1
- EBI3
- EBLN1
- EBNA1BP2
- ECEL1
- ECT2L
- EDARADD
- EDC3
- EDF1
- EDIL3
- EEF1A2
- EEF1D
- EEF1E1
- EEF2
- EFCAB2
- EFCAB6
- EFCBP2
- EFCC1
- EFEMP1
- EFEMP2
- EFHC1
- EFHD2
- EFTUD2
- EGLN2
- EGLN3
- EGR3
- EGR4
- EHD1
- EHMT1
- EI24
- EIF1B
- EIF2A
- EIF2B1
- EIF2B2
- EIF2B3
- EIF2B4
- EIF2B5
- EIF2C2
- EIF2S2
- EIF3B
- EIF3C
- EIF3D
- EIF3EIP
- EIF3F
- EIF3G
- EIF3H
- EIF3I
- EIF3J
- EIF3K
- EIF3M
- EIF3S6
- EIF4A1
- EIF4A2
- EIF4B
- EIF4E1B
- EIF4E2
- EIF4E3
- EIF4EBP2
- EIF4ENIF1
- EIF4G2
- EIF4G3
- EIF5
- EIF5A
- EIF5A2
- EIF5B
- ELF1
- ELF2
- ELK3
- ELK4
- ELL3
- ELMO1
- ELMO2
- ELOVL4
- ELOVL5
- ELP2
- ELP3
- EMCN
- EMG1
- EMILIN1
- EML1
- EML4
- EMP1
- EMP2
- EMP3
- EMSY
- EMX2
- ENKD1
- EP400
- EPB41L1
- EPB41L2
- EPB41L3
- EPB41L5
- EPB49
- EPC1
- EPHA10
- EPHA6
- EPHA7
- EPHA8
- EPHB3
- EPHB6
- EPN1
- EPN2
- EPS15L1
- EQTN
- ERAF
- ERAP2
- ERC1
- ERCC1
- ERG28
- ERGIC3
- ERLEC1
- ERLIN2
- ERMAP
- ERO1L
- ERO1LB
- ERRFI1
- ERV3
- ESCO1
- ESF1
- ESM1
- ETFA
- ETFB
- ETHE1
- ETV1
- ETV7
- EVA1A
- EVI2B
- EVI5
- EXOC1
- EXOC2
- EXOC3
- EXOC3L
- EXOC4
- EXOC5
- EXOC6
- EXOC7
- EXOC8
- EXT1
- EXTL1
- EXTL2
- EXTL3
- EYA1
- EYA2
- EYA4
- Enah/Vasp like
- Enkurin
- Envoplakin
- Ezrin
- FA2H
- FABP2
- FABP9
- FAF1
- FAM105B
- FAM107A
- FAM114A2
- FAM118B
- FAM120A
- FAM129A
- FAM132A
- FAM134C
- FAM136A
- FAM13C
- FAM155A
- FAM172A
- FAM181A
- FAM198B
- FAM20A
- FAM20B
- FAM20C
- FAM32A
- FAM3B
- FAM3C
- FAM46A
- FAM47E STBD1
- FAM49A
- FAM57B
- FAM60A
- FAM62A
- FAM62B
- FAM76B
- FANCE
- FANCI
- FARP1
- FARP2
- FASTKD3
- FAT4
- FBLIM1
- FBLN2
- FBLN5
- FBP1
- FBRS
- FBXL4
- FBXL5
- FBXL7
- FBXO11
- FBXO2
- FBXO24
- FBXO28
- FBXO31
- FBXO32
- FBXO4
- FBXO5
- FBXO6
- FBXO7
- FBXO9
- FBXW2
- FBXW4
- FBXW5
- FBXW7
- FBXW8
- FCER1A
- FCER1G
- FCF1
- FCHO1
- FCHO2
- FCHSD1
- FCHSD2
- FCMR
- FCN1
- FCN2
- FCN3
- FCRL1
- FCRL2
- FCRL3
- FCRL4
- FCRL5
- FCRLA
- FER1L3
- FERMT1
- FERMT2
- FEZ1
- FEZ2
- FEZF1
- FGD2
- FGD3
- FGD4
- FGF10
- FGF12
- FGF14
- FGF17
- FGF3
- FGF6
- FGF7
- FGFBP1
- FGL1
- FHL3
- FHL5
- FHOD1
- FIBP
- FIGLA
- FILIP1
- FJX1
- FKBP10
- FKBP2
- FKBP3
- FKBP8
- FKBP9
- FKBPL
- FLII
- FLOT1
- FLRT1
- FLRT2
- FLRT3
- FLT4
- FLVCR1
- FLVCR2
- FMNL1
- FMNL2
- FMNL3
- FNBP1
- FNBP1L
- FNBP4
- FNDC3A
- FNIP1
- FOLR2
- FOSB
- FOSL1
- FOSL2
- FOXD4
- FOXE3
- FOXF1
- FOXG1
- FOXH1
- FOXI1
- FOXJ2
- FOXK1
- FOXK2
- FOXM1
- FOXN1
- FOXN3
- FOXO6
- FOXP4
- FOXQ1
- FOXS1
- FRA10AC1
- FRAT1
- FRAT2
- FREM1
- FREM2
- FRMD6
- FRS3
- FSCN1
- FSCN2
- FSCN3
- FSTL3
- FXR1
- FXR2
- FXYD1
- FXYD3
- FXYD5
- FYB
- FZR1
- Fetuin B
- Fibromodulin
- GABARAPL2
- GABPA
- GABPB2
- GAD1
- GADD45B
- GADD45GIP1
- GAPVD1
- GAS1
- GAS2
- GAS2L1
- GAS7
- GATA5
- GATAD2B
- GBF1
- GBP1
- GBP5
- GBX2
- GCAT
- GCC1
- GCC2
- GCFC2
- GCHFR
- GCM1
- GCM2
- GCN1L1
- GDAP1
- GDF1
- GDF10
- GDF3
- GDF5
- GDF6
- GDF7
- GDI2
- GEMIN8
- GET4
- GFER
- GFI1
- GFI1B
- GFRA3
- GFRA4
- GFRAL
- GGA3
- GINS1
- GINS2
- GIPC2
- GJA10
- GJA3
- GJA4
- GJA5
- GJA8
- GJB2
- GJB4
- GJB5
- GJB7
- GJC1
- GJC2
- GJC3
- GJD2
- GJD3
- GJD4
- GKN1
- GLB1L3
- GLCCI1
- GLE1L
- GLG1
- GLMN
- GLRX
- GLRX3
- GLRX5
- GLS2
- GLTP
- GLTSCR2
- GMCL1
- GMEB1
- GMEB2
- GNA11
- GNA12
- GNAL
- GNAO1
- GNAT1
- GNAT2
- GNAT3
- GNAZ
- GNB1
- GNB2
- GNB3
- GNB4
- GNB5
- GNG11
- GNG12
- GNG13
- GNG2
- GNG3
- GNG4
- GNG5
- GNG7
- GNGT1
- GNGT2
- GNL2
- GNL3
- GNLY
- GNRH2
- GOLGA1
- GOLGA3
- GOLGA4
- GOLGA5
- GOLGA8A
- GOLGA8B
- GOLM1
- GOLT1B
- GON4L
- GOPC
- GORAB
- GOSR1
- GOSR2
- GPA33
- GPAA1
- GPATCH8
- GPBP1
- GPD1L
- GPHA2
- GPIHBP1
- GPM6A
- GPR107
- GPRIN2
- GPS1
- GPSM2
- GRAMD4
- GRAP
- GRASP65
- GREB1
- GREB1L
- GRID1
- GRIN3A
- GRIN3B
- GRINA
- GRINL1A
- GRIP2
- GRLF1
- GRWD1
- GRXCR1
- GSDMA
- GSDMB
- GSDMC
- GTF2E1
- GTF2E2
- GTF2F2
- GTF2H2
- GTF2H4
- GTF2H5
- GTF2IRD1
- GTF3C1
- GTF3C2
- GTF3C4
- GTF3C5
- GTPBP1
- GTPBP4
- GUF1
- GYPE
- GZMA
- GZMH
- GZMK
- GZMM
- Galectin 1
- Galectin 2
- Gamma synuclein
- Giantin
- Grancalcin
- HABP4
- HADHA
- HAPLN1
- HAPLN2
- HAUS1
- HAUS3
- HBP1
- HBXIP
- HCLS1
- HDLBP
- HEATR1
- HEBP1
- HEBP2
- HECTD4
- HECW1
- HECW2
- HEMGN
- HEMK1
- HEPACAM2
- HEPN1
- HERPUD1
- HES3
- HES5
- HES6
- HESX1
- HEXIM1
- HEXIM2
- HEY1
- HEY2
- HEYL
- HGFAC
- HHIP
- HIC1
- HIC2
- HID1
- HIF1AN
- HIF3A
- HINT1
- HIP1R
- HIP2
- HIRIP3
- HIST1H1A
- HIST1H1B
- HIST1H1C
- HIST1H1D
- HIST1H1E
- HIST1H1T
- HIST1H2AA
- HIST1H2AC
- HIST1H2AD
- HIST1H2AH
- HIST1H2AJ
- HIST1H2BA
- HIST1H2BB
- HIST1H2BD
- HIST1H2BH
- HIST1H2BJ
- HIST1H2BK
- HIST1H2BL
- HIST1H2BM
- HIST1H2BN
- HIST1H2BO
- HIST1H4G
- HIST1H4J
- HIST2H2AB
- HIST2H2AC
- HIST2H2BF
- HIST3H2A
- HIST3H2BB
- HIST3H3
- HIVEP1
- HIVEP2
- HIVEP3
- HKR1
- HLA DMA
- HLA DOA
- HLA DPB1
- HLA DQA2
- HLA DRB1
- HLA DRB5
- HM13
- HMBOX1
- HMCES
- HMG20A
- HMG20B
- HMGB2
- HMGN3
- HMGXB3
- HMHA1
- HN1L
- HNRNPA0
- HNRNPK
- HNRNPL
- HNRNPLL
- HNRNPR
- HNRPA3
- HNRPF
- HNRPH1
- HNRPH3
- HNRPU
- HNRPUL1
- HOMER2
- HOMER3
- HOOK1
- HOOK2
- HOOK3
- HOPX
- HORMAD1
- HORMAD2
- HOXA11
- HOXA13
- HOXA2
- HOXA3
- HOXA4
- HOXA5
- HOXA6
- HOXA7
- HOXB1
- HOXB13
- HOXB2
- HOXB3
- HOXB4
- HOXB5
- HOXB7
- HOXB8
- HOXB9
- HOXC10
- HOXC11
- HOXC12
- HOXC13
- HOXC4
- HOXC5
- HOXC6
- HOXC8
- HOXC9
- HOXD1
- HOXD10
- HOXD11
- HOXD12
- HOXD13
- HOXD3
- HOXD4
- HOXD8
- HOXD9
- HPS1
- HPS3
- HPS4
- HPS5
- HPS6
- HRBL
- HSD17B4
- HSF2
- HSF4
- HSPA14
- HSPA2
- HSPA4L
- HSPA6
- HSPB2
- HSPB3
- HSPB6
- HSPB8
- HSPBP1
- HSPH1
- HYLS1
- HYOU1
- ICAM5
- ID2
- IER3
- IER3IP1
- IFI16
- IFI27
- IFI35
- IFI6
- IFIT1
- IFIT2
- IFIT3
- IFNA10
- IFNA13
- IFNA16
- IFNA17
- IFNA21
- IFNA4
- IFNA5
- IFNA6
- IFNA8
- IFNAR1
- IFNB1
- IFNK
- IFT20
- IFT57
- IFT74
- IFT80
- IFT81
- IFT88
- IGF2BP1
- IGF2BP2
- IGF2BP3
- IGFALS
- IGFBP1
- IGFBP2
- IGFBP4
- IGFBP5
- IGFBP6
- IGFLR1
- IGHMBP2
- IKZF2
- IKZF3
- IKZF4
- IL12A
- IL17RD
- IL18BP
- IL1F10
- IL1RAP
- IL1RL1
- IL1RL2
- IL22RA1
- IL31RA
- IL36A
- IL36B
- IL36G
- ILF2
- IMMT
- IMP3
- IMP4
- IMPG1
- INADL
- INF2
- ING2
- ING3
- ING4
- INHA
- INHBC
- INPP4B
- INPP5A
- INPP5F
- INSL4
- INSL5
- INSM1
- INSRR
- INTS1
- INTS11
- INTS12
- INTS3
- INTS6
- INTS7
- INTS8
- INTS9
- INVS
- IPO11
- IPO13
- IPO4
- IPO5
- IPO7
- IPO8
- IPO9
- IQCB1
- IQCE
- IQSEC1
- IRF2
- IRF2BPL
- IRF5
- IRX2
- IRX3
- IRX4
- IRX5
- IRX6
- ISG15
- ISG20
- ITGB1BP1
- ITGB3BP
- ITIH1
- ITIH2
- ITIH3
- ITM2B
- ITM2C
- ITPA
- IVNS1ABP
- IWS1
- IZUMO1
- Intelectin 2
- JAG2
- JAKMIP2
- JAKMIP3
- JAM3
- JAZF1
- JKAMP
- JMJD1C
- JPH1
- JPH2
- JPH3
- JPH4
- JUNB
- KAAG1
- KANSL3
- KATNB1
- KATNBL1
- KBTBD10
- KBTBD7
- KCMF1
- KCNH1
- KCNH5
- KCNIP1
- KCNIP4
- KCNK5
- KCNRG
- KCNU1
- KCTD12
- KCTD13
- KCTD15
- KCTD8
- KDELR2
- KDELR3
- KDM1B
- KDM2B
- KDM3A
- KDM6B
- KDM7A
- KDM8
- KHDRBS3
- KHSRP
- KIAA0101
- KIAA0157
- KIAA0174
- KIAA0182
- KIAA0319
- KIAA0368
- KIAA0409
- KIAA0430
- KIAA0460
- KIAA0930
- KIAA0999
- KIAA1012
- KIAA1219
- KIAA1279
- KIAA1377
- KIAA1530
- KIAA1539
- KIAA1683
- KIAA1797
- KIAA1826
- KIAA1949
- KIAA1967
- KIF13A
- KIF14
- KIF15
- KIF16B
- KIF17
- KIF18A
- KIF1A
- KIF1C
- KIF20A
- KIF21A
- KIF22
- KIF24
- KIF2A
- KIF2C
- KIF3A
- KIF3C
- KIF5A
- KIF5B
- KIF6
- KIFAP3
- KIFC3
- KIN
- KIRREL
- KIRREL2
- KLC3
- KLC4
- KLF10
- KLF11
- KLF12
- KLF14
- KLF17
- KLF5
- KLF6
- KLF7
- KLHDC3
- KLK1
- KLK10
- KLK11
- KLK12
- KLK14
- KLK15
- KLK2
- KLRG1
- KMT2E
- KPNA3
- KPNA4
- KPNA5
- KPNA6
- KRBA2
- KREMEN1
- KRR1
- KRT23
- KRT24
- KRT25
- KRT27
- KRT28
- KRT31
- KRT32
- KRT33A
- KRT33B
- KRT35
- KRT36
- KRT37
- KRT38
- KRT39
- KRT71
- KRT72
- KRT73
- KRT74
- KRT75
- KRT77
- KRT78
- KRT79
- KRT80
- KRT81
- KRT83
- KRT84
- KRT85
- KRTDAP
- KTN1
- Kalirin
- Kallistatin
- Keratocan
- Kiaa1024
- Kiaa1107
- Kiaa1755
- LACTB2
- LAIR2
- LAMTOR4
- LANCL1
- LANCL2
- LAPTM4A
- LARP1
- LARP6
- LASP1
- LAT2
- LBH
- LBX1
- LCA5
- LCOR
- LCP1
- LCTL
- LDB1
- LDB2
- LDLRAP1
- LDOC1L
- LECT1
- LEFTY2
- LEKTI
- LETM1
- LGALS13
- LGALS3BP
- LGI2
- LGMN
- LGP2
- LGTN
- LHFP
- LHFPL2
- LHX2
- LHX3
- LHX4
- LHX6
- LILRA1
- LIM2
- LIMA1
- LIMCH1
- LIMD1
- LIME1
- LIMS2
- LIN7A
- LIN7B
- LIN7C
- LIN9
- LINGO2
- LITAF
- LLGL1
- LMAN1
- LMAN2
- LMAN2L
- LMBR1
- LMBR1L
- LMBRD1
- LMLN
- LMO1
- LMO4
- LMO7
- LMOD3
- LMTK3
- LMX1A
- LMX1B
- LOC124220
- LOC642658
- LOXHD1
- LPCAT1
- LPIN1
- LPXN
- LRBA
- LRCH4
- LRDD
- LRG1
- LRIT3
- LRP10
- LRP11
- LRP1B
- LRP3
- LRP6
- LRPPRC
- LRRC16A
- LRRC17
- LRRC23
- LRRC39
- LRRC4
- LRRC41
- LRRC48
- LRRC50
- LRRC8A
- LRRC8D
- LRRC8E
- LRRFIP1
- LRRN1
- LRRN2
- LRTOMT
- LSM1
- LSM10
- LSM4
- LSM5
- LSM6
- LSM7
- LSM8
- LSP1
- LST1
- LTBP2
- LTBP3
- LTN1
- LUC7L
- LUC7L2
- LUC7L3
- LY6E
- LYAR
- LYK5
- LYL1
- LYPD1
- LYPD3
- LYPD8
- LYSMD3
- LZTR1
- LZTS1
- LZTS3
- Layilin
- Liprin alpha 1
- Loricrin
- MAATS1
- MAB21L2
- MAD2L1BP
- MAD2L2
- MADCAM1
- MAF1
- MAGOH
- MAK16
- MALL
- MAML2
- MAP1A
- MAP1B
- MAP1LC3A
- MAP1S
- MAP3K19
- MAP4K3
- MAP6
- MAP7
- MAPK1IP1L
- MAPK8IP2
- MAPKAP1
- MAPRE1
- MAPRE2
- MAPRE3
- MARCKSL1
- MARK2
- MARVELD2
- MB21D1
- MBNL1
- MBNL2
- MCF2L
- MCFD2
- MCL1
- MCM10
- MCM3AP
- MCM5
- MCM8
- MCRS1
- MDFI
- MDM4
- MDN1
- MEAF6
- MED10
- MED11
- MED12L
- MED13
- MED18
- MED19
- MED4
- MED9
- MEF2C
- MEGF10
- MEI1
- MEIS1
- MEIS2
- MEMO1
- MEOX1
- MEOX2
- MEP1B
- MEPE
- MESDC2
- MESP1
- MESP2
- METRN
- METTL14
- METTL16
- METTL24
- MEX3B
- MEX3D
- MFAP1
- MFAP2
- MFAP5
- MFGE8
- MFSD2
- MFSD7
- MFSD8
- MGEA5
- MIA2
- MICAL1
- MICAL2
- MIER1
- MIGA1
- MIS12
- MIS18A
- MIS18BP1
- MKI67IP
- MKKS
- MKL1
- MKL2
- MKRN3
- MKX
- MLANA
- MLC1
- MLF1
- MLF1IP
- MLLT1
- MLLT10
- MLLT3
- MLST8
- MMAA
- MMACHC
- MMEL1
- MMP8
- MMS19
- MMS22L
- MNDA
- MNX1
- MOAP1
- MOBKL1A
- MOCS1
- MOCS2
- MOGAT3
- MON2
- MORC2
- MORC3
- MOSC2
- MPDU1
- MPDZ
- MPHOSPH1
- MPHOSPH10
- MPLKIP
- MPP2
- MPP6
- MPV17
- MPZL1
- MRAP2
- MRCL3
- MRFAP1
- MROH8
- MRPL1
- MRPL10
- MRPL11
- MRPL12
- MRPL15
- MRPL17
- MRPL18
- MRPL19
- MRPL20
- MRPL22
- MRPL23
- MRPL24
- MRPL28
- MRPL3
- MRPL30
- MRPL32
- MRPL33
- MRPL37
- MRPL39
- MRPL4
- MRPL40
- MRPL55
- MRPS11
- MRPS12
- MRPS16
- MRPS17
- MRPS18A
- MRPS18B
- MRPS2
- MRPS21
- MRPS22
- MRPS24
- MRPS25
- MRPS26
- MRPS28
- MRPS30
- MRPS33
- MRPS35
- MRPS5
- MRPS6
- MRPS7
- MRPS9
- MRS2L
- MRVI1
- MS4A2
- MS4A3
- MS4A7
- MSI1
- MSMO1
- MST1
- MST1R
- MT1B
- MT1E
- MT1F
- MT1X
- MTCH2
- MTERF
- MTF1
- MTFMT
- MTFR1L
- MTIF2
- MTMR12
- MTMR14
- MTMR2
- MTMR3
- MTMR6
- MTMR9
- MTO1
- MTPN
- MTRF1L
- MTRNR2L6
- MTRNR2L8
- MTSS1
- MTX1
- MUCL1
- MUPCDH
- MX2
- MXD4
- MXI1
- MYADM
- MYBBP1A
- MYBPC1
- MYBPC2
- MYCL1
- MYF6
- MYH1
- MYH13
- MYH14
- MYH15
- MYH2
- MYH3
- MYH4
- MYH7B
- MYH8
- MYL1
- MYL6
- MYL6B
- MYL9
- MYLIP
- MYLPF
- MYO15A
- MYO18B
- MYO1A
- MYO1B
- MYO1C
- MYO1E
- MYO1F
- MYO3A
- MYO5B
- MYO6
- MYOZ1
- MYT1
- MYT1L
- MZB1
- MZF1
- Mammaglobin A
- Mammaglobin B
- Matrilin 1
- Matrilin 2
- Matrilin 3
- MeCP2
- Metallothionein 3
- Muskelin
- Myocardin
- Myocilin
- N myc interactor
- NAA35
- NAB1
- NAB2
- NABP1
- NAGLU
- NANOS1
- NAP1L1
- NAP1L4
- NAPB
- NAPG
- NAPSA
- NAT6
- NAV1
- NBEAL2
- NBPF10
- NBPF16
- NCAPD2
- NCAPD3
- NCAPG
- NCAPG2
- NCAPH
- NCAPH2
- NCDN
- NCKAP1
- NCLN
- NCOA7
- NDC1
- NDE1
- NDEL1
- NDFIP1
- NDFIP2
- NDRG2
- NDRG4
- NDUFAF1
- NDUFAF4
- NECAP2
- NEFM
- NEGR1
- NEIL3
- NEK8
- NELL1
- NEO1
- NET1
- NETO1
- NEURL
- NEURL2
- NEUROD2
- NFASC
- NFATC1
- NFATC3
- NFATC4
- NFE2
- NFIA
- NFIL3
- NFIX
- NFKB2
- NFKBIB
- NFKBIE
- NFKBIZ
- NFRKB
- NFS1
- NFX1
- NFYA
- NFYB
- NFYC
- NHLH1
- NHLRC1
- NIF3L1
- NINJ1
- NIP30
- NIP7
- NIPA2
- NIPBL
- NISCH
- NKAIN1
- NKG7
- NKIRAS2
- NKTR
- NKX2 2
- NKX2 3
- NKX3 2
- NKX6 1
- NKX6 2
- NLE1
- NLGN1
- NLGN2
- NLRC4
- NLRP1
- NLRP12
- NLRP2
- NLRP4
- NLRP7
- NME4
- NME8
- NOA1
- NOB1
- NOBOX
- NOC2L
- NOD1
- NOL1
- NOL12
- NOL4
- NOL5A
- NOL6
- NOL7
- NOL8
- NOL9
- NOLA2
- NOM1
- NOP58
- NOS1AP
- NOVA1
- NOX5
- NPDC1
- NPHP4
- NPLOC4
- NPM3
- NPRL2
- NPRL3
- NPTN
- NPTX1
- NPTX2
- NPW
- NRARP
- NRBP1
- NRCAM
- NRD1
- NREP
- NRIP2
- NRN1
- NRSN1
- NRSN2
- NRXN1
- NRXN2
- NRXN3
- NSL1
- NSMCE4A
- NSUN2
- NT5C1A
- NUB1
- NUCKS1
- NUDC
- NUDCD2
- NUDCD3
- NUDT17
- NUDT6
- NUF2
- NUFIP1
- NUFIP2
- NUMBL
- NUPL1
- NUPL2
- NUPR1
- NXPH1
- NXPH3
- NXT1
- Nephronectin
- Neurocan
- Neurogenin 1
- Neurogenin 2
- Neurotrimin
- Neurotrophin 4
- Ninein
- OBSL1
- ODF1
- ODF2
- OLIG2
- ONECUT1
- OPA3
- OPCML
- OR10K1
- OR2G6
- OR4F5
- OR6C75
- ORC3
- ORC4
- ORC5
- ORC6
- ORM1
- OSBP2
- OSBPL11
- OSBPL1A
- OSBPL2
- OSBPL3
- OSBPL5
- OSBPL8
- OSTF1
- OSTM1
- OSTalpha
- OSTbeta
- OTOF
- OTOR
- OTUD1
- OTUD4
- OTUD6B
- OTUD7B
- OTX1
- OVGP1
- OXR1
- Opticin
- Osteomodulin
- Otogelin
- PA2G4
- PABPC3
- PABPN1
- PACRG
- PACS1
- PADI1
- PADI3
- PAG1
- PAIP2
- PALM
- PAM16
- PAN3
- PANK1
- PANX1
- PAPPA2
- PAQR5
- PAQR6
- PAQR7
- PAQR8
- PAQR9
- PARD3
- PARD3B
- PARD6A
- PARD6B
- PARL
- PARP16
- PARP2
- PARP3
- PARVA
- PARVB
- PARVG
- PATZ1
- PAX1
- PAX4
- PAXBP1
- PAXIP1
- PBRM1
- PBX2
- PBX3
- PCBP1
- PCBP2
- PCBP4
- PCDH1
- PCDH10
- PCDH12
- PCDH15
- PCDH17
- PCDH18
- PCDH20
- PCDH7
- PCDH8
- PCDHA2
- PCDHA3
- PCDHA4
- PCDHA5
- PCDHA6
- PCDHA9
- PCDHB11
- PCDHB12
- PCDHB13
- PCDHB14
- PCDHB15
- PCDHB16
- PCDHB2
- PCDHB3
- PCDHB4
- PCDHB5
- PCDHB7
- PCGF1
- PCGF2
- PCGF5
- PCGF6
- PCID2
- PCLKC
- PCLO
- PCNX
- PCOLCE2
- PCTK2
- PDAP1
- PDCD4
- PDCD5
- PDCD6IP
- PDCD7
- PDGFB
- PDGFD
- PDIA2
- PDLIM1
- PDLIM2
- PDLIM4
- PDLIM7
- PDPR
- PDS5A
- PDS5B
- PDSS2
- PDZD2
- PDZK1IP1
- PDZRN3
- PEA15
- PEG10
- PELI1
- PELI2
- PEN 2
- PERP
- PES1
- PEX1
- PEX10
- PEX11A
- PEX11B
- PEX12
- PEX13
- PEX3
- PFDN1
- PFDN2
- PFDN4
- PFDN5
- PFN2
- PGA5
- PGAP1
- PGDS
- PGRMC2
- PHAX
- PHC1
- PHC2
- PHC3
- PHF1
- PHF10
- PHF12
- PHF2
- PHF20
- PHF21A
- PHF3
- PHLDA1
- PHLDA2
- PHOX2A
- PHOX2B
- PHTF2
- PHYHIP
- PIBF1
- PICALM
- PIGF
- PIGT
- PIGW
- PIK3C2G
- PILRA
- PILRB
- PIP4K2C
- PITPNB
- PITPNM1
- PITX1
- PITX3
- PIWIL1
- PKD1L1
- PKD2L1
- PKIA
- PKIB
- PKIG
- PKNOX1
- PLAG1
- PLAGL1
- PLAGL2
- PLD5
- PLDN
- PLEK2
- PLEKHA1
- PLEKHA5
- PLEKHA6
- PLEKHA8
- PLEKHB2
- PLEKHF2
- PLEKHG4
- PLEKHG5
- PLEKHM1
- PLEKHO1
- PLRG1
- PLSCR2
- PLSCR4
- PLSCR5
- PLVAP
- PLXDC1
- PLXDC2
- PLXNA2
- PLXNB1
- PLXNB2
- PLXND1
- PM20D1
- PMFBP1
- PMP2
- PMS1
- PNISR
- PNMA8A
- PNO1
- PNRC1
- PNRC2
- PODXL2
- POGZ
- POLD4
- POLE4
- POLG2
- POLR1C
- POLR1D
- POLR2E
- POLR2F
- POLR2H
- POLR2K
- POLR2L
- POLR3G
- POM121
- POMP
- POPDC2
- POPDC3
- POT1
- POU2AF1
- POU2F3
- POU3F1
- POU3F2
- POU4F2
- POU4F3
- PPARGC1B
- PPFIA4
- PPFIBP1
- PPHLN1
- PPIP5K2
- PPP1R13B
- PPP1R13L
- PPP1R14A
- PPP1R17
- PPP1R27
- PPP1R3C
- PPP1R3G
- PPP1R9A
- PPP1R9B
- PPP3CA
- PPP3R2
- PPRC1
- PRAM1
- PRAP1
- PRB1
- PRB3
- PRCD
- PRDM2
- PREB
- PRELP
- PREX1
- PREX2
- PRICKLE1
- PRICKLE4
- PRIMA1
- PRKAA2
- PRKCB1
- PRKCH
- PRKRA
- PRND
- PRNP
- PROP1
- PRPF19
- PRPF3
- PRPF4
- PRPF40A
- PRPF40B
- PRPF8
- PRR29
- PRR35
- PRR4
- PRR9
- PRRT2
- PRRX2
- PRSS2
- PRSS3
- PRSS55
- PRSS8
- PRUNE2
- PSAPL1
- PSCDBP
- PSD3
- PSEN2
- PSG3
- PSG5
- PSG6
- PSG9
- PSME1
- PSME2
- PSME3
- PSME4
- PSMF1
- PSMG1
- PSRC1
- PSTPIP2
- PTBP2
- PTCH2
- PTF1A
- PTK2B
- PTOV1
- PTPMT1
- PTPRN2
- PTRF
- PTTG1IP
- PUF60
- PUM1
- PUM3
- PURB
- PURG
- PVRIG
- PWP1
- PWP2
- PWWP2A
- PXDN
- PXMP4
- PYCARD
- PYCR2
- PYGO1
- PYGO2
- Peflin
- Periplakin
- Phosducin
- Phosducin like
- Plakophilin 3
- Plakophilin 4
- QKI
- QRFP
- RAB10
- RAB11FIP1
- RAB11FIP2
- RAB11FIP3
- RAB11FIP4
- RAB13
- RAB14
- RAB15
- RAB17
- RAB18
- RAB1B
- RAB21
- RAB22A
- RAB23
- RAB25
- RAB26
- RAB2A
- RAB30
- RAB31
- RAB33B
- RAB34
- RAB35
- RAB36
- RAB37
- RAB38
- RAB3A
- RAB3B
- RAB3C
- RAB3D
- RAB3IP
- RAB5B
- RAB5C
- RAB6A
- RAB6B
- RAB6C
- RAB7B
- RAB7L1
- RAB8A
- RAB8B
- RABAC1
- RABEPK
- RABGAP1
- RABIF
- RAD21
- RAD23A
- RAD51L1
- RAD51L3
- RAD54B
- RADIL
- RAE1
- RAG1
- RAG2
- RAI14
- RALBP1
- RALGPS1
- RALY
- RALYL
- RAMP2
- RANBP3
- RANBP9
- RAP1A
- RAP1B
- RAP1GAP2
- RAP2A
- RAP2B
- RAPGEF5
- RAPH1
- RARRES1
- RARRES3
- RASA4B
- RASAL2
- RASAL3
- RASD2
- RASGEF1A
- RASGRF1
- RASGRF2
- RASGRP1
- RASGRP2
- RASGRP3
- RASGRP4
- RASL11B
- RASSF1
- RASSF2
- RASSF8
- RB1CC1
- RBBP5
- RBBP6
- RBCK1
- RBFOX1
- RBM11
- RBM14
- RBM15
- RBM17
- RBM19
- RBM22
- RBM23
- RBM25
- RBM26
- RBM28
- RBM33
- RBM34
- RBM39
- RBM47
- RBM5
- RBM6
- RBM7
- RBM8A
- RBM9
- RBMS1
- RBP1
- RBP2
- RBP7
- RBPMS
- RC3H1
- RCAN2
- RCAN3
- RCBTB2
- RCC2
- RCHY1
- RCOR1
- RDBP
- RDH13
- REC8
- REEP1
- REEP2
- REEP5
- REG1A
- REG1B
- REG3A
- REG3G
- REG4
- REL
- RELB
- RELT
- REPS1
- RERE
- RERG
- RETNLB
- RETSAT
- REV1
- RFT1
- RFX2
- RFX3
- RFX4
- RFX5
- RFX6
- RFXANK
- RFXAP
- RGL2
- RGPD5
- RGS1
- RGS10
- RGS11
- RGS12
- RGS13
- RGS14
- RGS17
- RGS18
- RGS20
- RGS3
- RGS5
- RGS6
- RGS7
- RGS8
- RGS9BP
- RHBDF1
- RHBDF2
- RHOB
- RHOBTB1
- RHOBTB2
- RHOBTB3
- RHOJ
- RHOQ
- RIC8A
- RIF1
- RIMBP2
- RIN1
- RINT1
- RIOX1
- RIOX2
- RIPOR2
- RIT1
- RIT2
- RMI1
- RNASE6
- RNASEH2C
- RNF11
- RNF122
- RNF13
- RNF135
- RNF139
- RNF146
- RNF183
- RNF213
- RNF220
- RNF24
- RNF26
- RNF32
- RNF38
- RNF43
- RNF7
- RNGTT
- ROBO2
- ROBO3
- ROBO4
- ROCK2
- ROM1
- RP1
- RP9
- RPAIN
- RPGRIP1
- RPH3AL
- RPLP0
- RPS27A
- RRAD
- RRAGA
- RRAGC
- RRAS
- RRAS2
- RREB1
- RRP1
- RRP12
- RRP15
- RRP9
- RRS1
- RSF1
- RSL1D1
- RSPH1
- RSPH10B
- RSPH14
- RSPH3
- RSPH4A
- RSPH6A
- RSPH9
- RSPO2
- RSPO3
- RSU1
- RTFDC1
- RTKN
- RTKN2
- RTN1
- RTN2
- RTN3
- RTRAF
- RUFY1
- RUNDC3A
- RWDD2B
- RWDD3
- RYBP
- RYR3
- Radixin
- SAA2
- SAC3D1
- SAE1
- SAFB2
- SALL2
- SALL3
- SAMD9
- SAMM50
- SAR1B
- SARNP
- SART1
- SART3
- SASH1
- SASS6
- SATB1
- SBF1
- SBF2
- SBNO2
- SC65
- SCAMP1
- SCAMP2
- SCAMP3
- SCAMP5
- SCAND1
- SCFD1
- SCG3
- SCG5
- SCGB1D2
- SCGB3A1
- SCGB3A2
- SCIN
- SCMH1
- SCRN1
- SCUBE1
- SCYL2
- SCYL3
- SDAD1
- SDCCAG8
- SDF4
- SDHAF2
- SDK1
- SDK2
- SDPR
- SEC13
- SEC14L1
- SEC14L2
- SEC16A
- SEC22A
- SEC23A
- SEC23B
- SEC23IP
- SEC24C
- SEC24D
- SEC31A
- SEC62
- SEC63
- SECISBP2
- SECTM1
- SEL1L
- SELENBP1
- SELT
- SEMA3B
- SEMA3C
- SEMA3F
- SEMA4A
- SEMA4B
- SEMA4C
- SEMA4F
- SEMA4G
- SEMA5A
- SEMA6A
- SEMA6C
- SEMG2
- SENP3
- SEPT1
- SEPT10
- SEPT11
- SEPT12
- SEPT14
- SEPT2
- SEPT3
- SEPT4
- SEPT5
- SEPT7
- SERAC1
- SERBP1
- SERINC1
- SERINC3
- SERP1
- SERPINA9
- SERPINB1
- SERPINB10
- SERPINB3
- SERPINB4
- SERPINB6
- SERPINB7
- SERPINB8
- SERPINB9
- SERPINE2
- SERPINI1
- SERPINI2
- SERTAD1
- SESN1
- SESN2
- SESN3
- SETBP1
- SETD5
- SETD6
- SEZ6
- SEZ6L
- SF3A1
- SF3A2
- SF3A3
- SF3B3
- SF3B4
- SF3B5
- SFI1
- SFMBT1
- SFRP2
- SFRP4
- SFRP5
- SFRS11
- SFRS12
- SFRS14
- SFRS16
- SFRS2
- SFRS2IP
- SFRS3
- SFRS4
- SFRS5
- SFRS6
- SFSWAP
- SFXN1
- SGCB
- SGCE
- SGCZ
- SGIP1
- SGSM1
- SGSM2
- SGTA
- SH2D1B
- SH2D2A
- SH2D3A
- SH2D3C
- SH2D4A
- SH3BGR
- SH3BGRL3
- SH3BP4
- SH3BP5
- SH3GL1
- SH3GL2
- SH3GL3
- SH3GLB1
- SH3GLB2
- SH3PXD2A
- SH3TC2
- SHANK2
- SHC2
- SHC3
- SHCBP1
- SHOC2
- SHROOM3
- SIDT1
- SIGIRR
- SIGLEC10
- SIGLEC9
- SIL1
- SIN3B
- SIPA1L1
- SIPA1L2
- SIT1
- SIVA1
- SIX1
- SIX2
- SIX4
- SIX5
- SIX6OS1
- SKIL
- SKIV2L2
- SLA2
- SLAMF6
- SLAMF7
- SLAMF8
- SLC12A8
- SLC12A9
- SLC13A2
- SLC13A5
- SLC15A4
- SLC16A5
- SLC17A3
- SLC17A8
- SLC22A13
- SLC22A24
- SLC22A8
- SLC25A16
- SLC25A21
- SLC25A24
- SLC26A8
- SLC27A6
- SLC2A11
- SLC2A13
- SLC2A14
- SLC2A4RG
- SLC2A7
- SLC2A9
- SLC30A1
- SLC30A3
- SLC33A1
- SLC35C2
- SLC36A2
- SLC37A2
- SLC38A1
- SLC38A10
- SLC39A10
- SLC39A12
- SLC41A3
- SLC45A3
- SLC47A1
- SLC47A2
- SLC50A1
- SLC5A4
- SLC6A15
- SLC6A18
- SLC6A20
- SLC6A6
- SLC7A14
- SLC7A5
- SLC8B1
- SLC9A10
- SLC9A11
- SLC9B2
- SLCO2A1
- SLFN11
- SLFN12
- SLIT2
- SLIT3
- SLITRK1
- SLITRK5
- SLITRK6
- SLMAP
- SLU7
- SLURP1
- SLX4IP
- SMAP1
- SMARCA5
- SMARCAD1
- SMARCAL1
- SMARCC1
- SMARCC2
- SMARCD1
- SMARCD2
- SMARCD3
- SMC2
- SMC4
- SMC5
- SMC6
- SMCP
- SMCR7L
- SMG5
- SMG7
- SMIM20
- SMOC2
- SMTN
- SMYD4
- SNAG1
- SNAP91
- SNAPAP
- SNAPC1
- SNAPC2
- SNAPC3
- SNAPC5
- SNCAIP
- SNF8
- SNIP1
- SNRPA1
- SNRPB
- SNRPB2
- SNRPD3
- SNRPG
- SNTB1
- SNTB2
- SNTG1
- SNX1
- SNX17
- SNX2
- SNX21
- SNX3
- SNX4
- SNX5
- SNX9
- SOCS2
- SOCS4
- SOCS6
- SOCS7
- SOGA1
- SOGA3
- SORBS3
- SORL1
- SOSTDC1
- SOX11
- SOX12
- SOX13
- SOX14
- SOX15
- SOX17
- SOX18
- SOX21
- SOX4
- SOX5
- SOX6
- SOX7
- SOX8
- SP110
- SPA17
- SPACA3
- SPATA2
- SPATA5
- SPATA7
- SPATC1L
- SPATS2L
- SPC24
- SPDEF
- SPDYE1
- SPECC1
- SPEF1
- SPEF2
- SPEN
- SPG20
- SPG21
- SPHK2
- SPI1
- SPIB
- SPIN1
- SPINK1
- SPINK2
- SPINLW1
- SPINT1
- SPN1
- SPOCK1
- SPOCK2
- SPOP
- SPRED2
- SPRED3
- SPRR1A
- SPRR1B
- SPRR2A
- SPRR3
- SPRTN
- SPRY1
- SPRY4
- SPRYD7
- SPSB1
- SPTB
- SPTBN2
- SPTBN4
- SPTBN5
- SPTLC2
- SRGAP2
- SRPRB
- SRRM1
- SRRM2
- SS18L1
- SSBP1
- SSBP2
- SSBP3
- SSC4D
- SSFA2
- SSR1
- SSR2
- SSX2IP
- ST3GAL3
- ST7
- ST8SIA6
- STAB1
- STAB2
- STAG1
- STAM2
- STAMBP
- STAP1
- STAP2
- STAT5B
- STATH
- STAU1
- STAU2
- STC2
- STCH
- STEAP4
- STIL
- STIM1
- STK10
- STK17A
- STK35L1
- STMN2
- STOML1
- STOML2
- STOX1
- STRBP
- STRN
- STRN3
- STRN4
- STX12
- STX17
- STX1B
- STX2
- STX5
- STX7
- STXBP1
- STXBP5
- STYXL1
- SUB1
- SUFU
- SUGT1
- SULT1A3
- SUMO3
- SUPT16H
- SUPT20H
- SUPT6H
- SUPT7L
- SURF1
- SURF4
- SURF6
- SV2B
- SVIL
- SWAP70
- SYCP1
- SYCP3
- SYN3
- SYNCRIP
- SYNE2
- SYNGR1
- SYNJ2
- SYNPR
- SYPL1
- SYT11
- SYT13
- SYT14
- SYT2
- SYT5
- SYT6
- SYT7
- SYT9
- SZT2
- Sacsin
- Sarcolipin
- Secretagogin
- Serglycin
- Serpina3f
- Stratifin
- Syndecan 2
- Syndecan 3
- Syntaphilin
- Syntenin 1
- Syntenin 2
- TAAR1
- TAC3
- TAC4
- TACC2
- TACC3
- TACSTD2
- TADA3L
- TAF12
- TAF13
- TAF15
- TAF1D
- TAF2
- TAF3
- TAF4B
- TAF6
- TAF8
- TAGLN3
- TAL1
- TAL2
- TANGO6
- TARBP2
- TAS2R42
- TAX1BP1
- TBC1D1
- TBC1D10A
- TBC1D15
- TBC1D24
- TBC1D3
- TBC1D4
- TBC1D9
- TBCA
- TBCB
- TBCD
- TBCE
- TBL1XR1
- TBL3
- TBPL1
- TBRG1
- TBX19
- TBX21
- TBX3
- TBX6
- TC2N
- TCEA1
- TCEA2
- TCEB3
- TCF12
- TCF15
- TCF19
- TCF20
- TCF4
- TCF7
- TCHP
- TCIM
- TCL1A
- TCP10L
- TCP11
- TCTEX1D4
- TCTN3
- TDP1
- TDRD3
- TDRD7
- TDRKH
- TEAD3
- TEAD4
- TECTA
- TECTB
- TEGT
- TEKT1
- TELO2
- TERF2IP
- TEX10
- TEX19
- TEX35
- TFAM
- TFAP2B
- TFAP2C
- TFAP4
- TFDP2
- TFEB
- TFEC
- TFIP11
- TFPT
- TGFBI
- TGIF2
- TGOLN2
- TH1L
- THAP2
- THAP6
- THAP7
- THOC1
- THRAP3
- THRSP
- THSD1
- THYN1
- TIAL1
- TIMD4
- TIMMDC1
- TIMP2
- TIMP3
- TINAG
- TINAGL1
- TINF2
- TINP1
- TIP39
- TIPIN
- TJAP1
- TJP3
- TLE2
- TLE3
- TLE4
- TLL1
- TLL2
- TLR10
- TLR6
- TLX
- TLX1
- TLX2
- TLX3
- TM4SF1
- TM4SF5
- TM9SF2
- TMBIM1
- TMBIM4
- TMC2
- TMC3
- TMC8
- TMCO1
- TMCO5A
- TMED1
- TMED10
- TMED2
- TMED3
- TMED4
- TMEFF2
- TMEM1
- TMEM10
- TMEM106B
- TMEM109
- TMEM117
- TMEM123
- TMEM130
- TMEM138
- TMEM150
- TMEM160
- TMEM176B
- TMEM18
- TMEM184B
- TMEM196
- TMEM200A
- TMEM215
- TMEM216
- TMEM219
- TMEM22
- TMEM242
- TMEM25
- TMEM26
- TMEM43
- TMEM44
- TMEM49
- TMEM51
- TMEM59
- TMEM66
- TMEM67
- TMEM9
- TMEPAI
- TMF1
- TMIGD1
- TMIGD2
- TMPRSS11F
- TMSB10
- TMTC2
- TMX1
- TMX4
- TNFAIP1
- TNFAIP8
- TNFAIP8L2
- TNFRSF21
- TNFSF18
- TNIP2
- TNNC2
- TNNI3K
- TNP1
- TNP2
- TNPO2
- TNRC15
- TNRC6A
- TNRC6B
- TNS1
- TNS4
- TOB1
- TOB2
- TOE1
- TOM1
- TOM1L1
- TOM1L2
- TOMM20
- TOMM22
- TOMM34
- TOMM40
- TOMM40L
- TOMM70A
- TONSL
- TOR1AIP1
- TOR2A
- TOX
- TP53I11
- TP53INP1
- TPD52
- TPD52L1
- TPD52L2
- TPM4
- TPPP
- TPT1 AS1
- TPX2
- TRA2A
- TRA2B
- TRAF3IP2
- TRAF4
- TRAK1
- TRAK2
- TRAM2
- TRANK1
- TRAP1
- TRAPPC3
- TRAPPC4
- TRAPPC9
- TRAT1
- TREM2
- TREML1
- TRERF1
- TRHDE
- TRIB3
- TRIM11
- TRIM22
- TRIM23
- TRIM25
- TRIM29
- TRIM3
- TRIM37
- TRIM38
- TRIM41
- TRIM55
- TRIM68
- TRIM69
- TRIM9
- TRIOBP
- TRIP10
- TRIP13
- TRIP4
- TRIP6
- TRMT12
- TRMT13
- TROAP
- TROVE2
- TSC22D1
- TSFM
- TSG 6
- TSHZ1
- TSHZ3
- TSPAN13
- TSPAN2
- TSPAN3
- TSPAN31
- TSPAN32
- TSPAN4
- TSPAN8
- TSPYL1
- TSSC1
- TTBK2
- TTC1
- TTC25
- TTC28
- TTC3
- TTC35
- TTC8
- TTF2
- TTI2
- TTLL3
- TTPAL
- TTRAP
- TUBA1B
- TUBA1C
- TUBA4A
- TUBB1
- TUBB2A
- TUBD1
- TUBE1
- TUBG1
- TUBG2
- TUBGCP2
- TUBGCP3
- TUBGCP4
- TUBGCP5
- TUBGCP6
- TUFM
- TULP1
- TUSC2
- TUSC3
- TWF1
- TWSG1
- TXLNB
- TXNDC12
- TXNDC5
- TXNDC9
- TXNL1
- TXNL4A
- TXNL4B
- TYSND1
- Tenascin R
- Thrombospondin 2
- Transgelin
- Translin
- Transportin 3
- Trichohyalin
- UACA
- UBAC1
- UBAP2
- UBAP2L
- UBASH3A
- UBE1C
- UBE1DC1
- UBE1L
- UBE1L2
- UBE2B
- UBE2C
- UBE2D2
- UBE2E1
- UBE2E2
- UBE2E3
- UBE2G1
- UBE2G2
- UBE2J1
- UBE2L6
- UBE2M
- UBE2N
- UBE2O
- UBE2R2
- UBE2V2
- UBE4A
- UBE4B
- UBIAD1
- UBL3
- UBL5
- UBL7
- UBP1
- UBTF
- UBXD2
- UBXD5
- UBXN6
- UCN2
- UCN3
- UEVLD
- UFD1L
- UFM1
- UHMK1
- UIMC1
- UNC13A
- UNC13B
- UNC13D
- UNC45A
- UNC5A
- UNC5C
- UNC5CL
- UNC84B
- UNC93A
- UPF1
- UPK3A
- UQCRC1
- UQCRC2
- UQCRFS1
- UQCRQ
- URI1
- USE1
- USF2
- USH1G
- USH2A
- USO1
- USP19
- USP22
- USP25
- USP39
- USP6NL
- UTF1
- UTP11L
- UTP15
- UTP18
- UTP3
- UTP6
- UVRAG
- Uteroglobin
- VAC14
- VAMP1
- VAMP4
- VANGL2
- VAPA
- VAPB
- VARS2
- VASH1
- VAT1
- VAT1L
- VAV2
- VAV3
- VAX1
- VCPIP1
- VEGFR1
- VIPAS39
- VNN2
- VPREB3
- VPS11
- VPS13A
- VPS13D
- VPS18
- VPS24
- VPS25
- VPS26B
- VPS28
- VPS29
- VPS33B
- VPS35
- VPS36
- VPS37A
- VPS39
- VPS45
- VPS4A
- VPS4B
- VPS52
- VPS53
- VPS72
- VSIG2
- VSX1
- VSX2
- VTA1
- VTCN1
- VTI1A
- VTI1B
- VWA2
- VWA5A
- WAPAL
- WBP1
- WBP2
- WBP4
- WDFY3
- WDHD1
- WDR1
- WDR24
- WDR26
- WDR3
- WDR33
- WDR36
- WDR37
- WDR45L
- WDR47
- WDR5
- WDR6
- WDR62
- WDR68
- WDR72
- WDR8
- WDR83
- WFDC1
- WFDC2
- WFDC5
- WFIKKN1
- WFIKKN2
- WFS1
- WIF1
- WIPF2
- WIPI1
- WIPI2
- WNT1
- WNT10A
- WNT10B
- WNT11
- WNT2B
- WNT3
- WNT3A
- WNT5B
- WNT7A
- WNT8A
- WNT8B
- WNT9A
- WNT9B
- WSB1
- WWC1
- WWTR1
- XAB2
- XAF1
- XBP1
- XIRP2
- XPO4
- XPO6
- XPOT
- YAF2
- YBX2
- YIF1A
- YIPF1
- YIPF3
- YIPF4
- YKT6
- YTHDC1
- YTHDC2
- YTHDF1
- YTHDF2
- YTHDF3
- YY1AP1
- ZBED3
- ZBED6
- ZBP1
- ZBTB1
- ZBTB11
- ZBTB17
- ZBTB20
- ZBTB32
- ZBTB40
- ZBTB48
- ZBTB7A
- ZBTB7B
- ZC3H12A
- ZC3H13
- ZC3HAV1
- ZCCHC17
- ZCCHC2
- ZCCHC6
- ZCCHC8
- ZDHHC14
- ZEB1
- ZFP106
- ZFP161
- ZFP36L1
- ZFP57
- ZFP64
- ZFP91
- ZFPM1
- ZFYVE1
- ZFYVE16
- ZFYVE19
- ZFYVE20
- ZFYVE26
- ZFYVE27
- ZFYVE28
- ZGPAT
- ZHX1
- ZHX2
- ZHX3
- ZIC1
- ZIC2
- ZKSCAN1
- ZKSCAN5
- ZMIZ1
- ZMIZ2
- ZMYM1
- ZMYM2
- ZMYND10
- ZMYND11
- ZNF10
- ZNF133
- ZNF143
- ZNF146
- ZNF16
- ZNF160
- ZNF19
- ZNF193
- ZNF202
- ZNF207
- ZNF217
- ZNF219
- ZNF224
- ZNF225
- ZNF23
- ZNF238
- ZNF239
- ZNF24
- ZNF259
- ZNF264
- ZNF267
- ZNF274
- ZNF277P
- ZNF281
- ZNF295
- ZNF3
- ZNF300
- ZNF318
- ZNF330
- ZNF331
- ZNF33A
- ZNF33B
- ZNF346
- ZNF35
- ZNF350
- ZNF364
- ZNF365
- ZNF37A
- ZNF383
- ZNF384
- ZNF408
- ZNF423
- ZNF43
- ZNF436
- ZNF44
- ZNF451
- ZNF452
- ZNF469
- ZNF471
- ZNF473
- ZNF497
- ZNF571
- ZNF593
- ZNF598
- ZNF638
- ZNF649
- ZNF655
- ZNF664
- ZNF676
- ZNF687
- ZNF7
- ZNF707
- ZNF71
- ZNF74
- ZNF76
- ZNF79
- ZNF8
- ZNF83
- ZP2
- ZP3
- ZP4
- ZRANB2
- ZSCAN21
- ZW10
- ZWINT
- ZYG11B
- ZZZ3
- ابيرجولين
- اديبونوترين
- اف بى ان 1
- ال سى ان 2
- البروتين البرومودومين 4
- البروتين المرتبط بالموت6
- البيومين المصل
- الفا تاكسلن
- الوحده الفرعيه بيتا للفولليتروبين
- امفيفيسين
- اميلوبلاستين
- انترلوكين 11
- انترلوكين 17ا
- انترلوكين 18
- انترلوكين 22
- انترلوكين 25
- انترلوكين 3
- انجيوجينين
- انزيم محول للانجيوتنسين 2
- انهدراز الكربونيك السادس
- ايه.ال. كيه 2
- بارن
- ببتيد منشط محلقة الادينيلات النخاميه
- براتشيورى
- بروبيردين
- بروتين 63 المتعلق بالتحول
- بروتين اصلاح الحمض النووى
- بروتين الزنك الاصبعى 684
- بروتين السربين اف 1
- بروتين المرتبط بتكون جينات الغضروف
- بروتين رابط للجلوبين المناعى
- بروتين زنك الاصبع 692
- بروتين شبيه ELAV 3
- بروتين كيناز سى ثيا
- بروتين معدل لنشاط المستقبل 1
- بروتين يوبيكويتينى قديم 1
- بروتيوغليكان 4
- بلميتول بروتين ثيوسترس 1
- بيريفيرين
- جالاكتوز ميوتاروتيز
- جزيء الالتصاق الخلوى 3
- جيجاكسونين
- جين XIAP
- داء البوليبات الغدى القولونى
- رابط لتفعيل الخلايا التائيه
- ستات3
- ستيرول 27 هيدروكسيلاز
- سموم عصبية يوزينيه
- سى اف ال ايه آر
- سيالوبروتين العظم
- سيتوكروم 2اي1
- سينابتوبودين 2
- سينابتوفيزين
- سينابسين I
- سينابسين II
- سينيمن
- صميم البروتين الشحمى بى
- عيلة انزيم الديهيدروجيناز 3
- عامل 6
- عامل النمو الفيبروبلاستى الاساسى
- عامل النمو المحول الفا
- عامل النمو المحول بيتا 1
- عامل النمو المحول بيتا 2
- عامل النمو المحول بيتا 3
- عامل النمو شبيه الانسولين 1
- عامل تثبيط هجرة البلاعم
- عامل نمو الخلايا الليفيه 21
- عامل نمو المشيمه
- عضو عيلة شيسا 4
- عنصر الاكسوسوم 3
- غاسدرمين د
- قناة الصوديوم Nav1.5
- كاثليسدسن
- كاثيبسين E
- كاسباز 1
- كتلة التمايز 154
- كتلة التمايز 209
- كتلة التمايز 278
- كرايوبايرين
- كولين استيل ترانسفيراز
- كيراتين 17
- كيراتين 20
- كيراتين 7
- كيراس
- كيموتربسين سى
- كيناز سيرين/ثريونين11
- كيناز ١ المرتبط بTANK
- لامك3
- لكتين نوع ج عيلة 5 عضو ا
- مبادل كالسيوم/بوتاسيوم/صوديوم 4
- مخلق لوكوتريين سي4
- مستضد الكريات البيضاء البشريه ا
- مستقبل 5 HT2A
- مستقبل ادينوسين A1
- مستقبل ادينوسين A2A
- مستقبل الاحماض الدهنيه الحره 1
- مستقبل الاستروجين بيتا
- مستقبل الانترلوكين 2
- مستقبل التاخيكينين 1
- مستقبل الثرومبوبويتين
- مستقبل الريانودين 2
- مستقبل السيكريتين
- مستقبل الكالسيتونين
- مستقبل انترلوكين 3 الفا
- مستقبل حمض الريتينويك الفا
- مستقبل حمض الريتينويك بيتا
- مستقبل حمض الريتينويك غاما
- مستقبل حمض الهيدروكسى كربوكسيل 1
- مستقبل دوبامين D1
- مستقبل دوبامين D2
- مستقبل دوبامين D4
- مستقبل دوبامين D5
- مستقبل ريتينويد اكس الفا
- مستقبل ريتينويد اكس بيتا
- مستقبل ريتينويد اكس جاما
- مستقبل عامل تحفيز المستعمرات 1
- مستقبل عامل نمو الخليه الليفيه 2
- مستقبل منشط لمكاثر البيروكسيسوم النوع الفا
- مستقبل منشط لمكاثر البيروكسيسوم النوع جاما
- مستقبل منشط لمكاثر البيروكسيسوم دلتا
- مستقبلات البروجسترون
- مضاد الفيروسات التى يسببها بروتين عبر الغشاء5
- معقد التوافق النسيجى الكبير ب
- مكون متمم 3
- مكون متمم 6
- مكون متمم 7
- موضع معقد GNAS
- مولد ضد الخليه اللمفاويه 75
- ميزوثيلين
- ميكروسيفالين
- ناقل احادى الامين الحويصلى 1
- ناقل الغشاء البلازمى احادى الامين
- ناقل النورابينفرين
- ناقل مشارك صوديوم/جلوكوز 2
- ناقلة سلفيد الجلوتاثيون مو 1
- نطاق الديسنتجرين والبروتيناز الفلزى المحتوى على البروتين 33
- نظام مستضد كيل
- هرمون ملوتن بيتا كتير الببتيد
- هيبسيدين
- هيدرولاز يوبكتين الكربوكسى الطرفى L1
- وحيدة بيتا لهرمونات البروتين السكرى
- چين
مصادر
تعديل- ↑ أ ب Hubbard، T. (1 يناير 2002). "The Ensembl genome database project". Nucleic Acids Research. ج. 30 ع. 1: 38–41. DOI:10.1093/nar/30.1.38. PMC:99161. PMID:11752248.
- ↑ "Ensembl 2011". Nucleic Acids Res. ج. 39 ع. Database issue: D800–D806. نوفمبر 2010. DOI:10.1093/nar/gkq1064. PMC:3013672. PMID:21045057.
- ↑ "Ensembl's 10th year". Nucleic Acids Res. ج. 38 ع. Database issue: D557–62. يناير 2010. DOI:10.1093/nar/gkp972. PMC:2808936. PMID:19906699.
- ↑ Davis، Charles Patrick (29 مارس 2021). "Medical definition of Genome Annotation". مؤرشف من الأصل في 2021-06-14. اطلع عليه بتاريخ 2022-08-07.
- ↑ Curwen، Val؛ Eyras، Eduardo؛ Andrews، T. Daniel؛ Clarke، Laura؛ Mongin، Emmanuel؛ Searle، Steven M. J.؛ Clamp، Michele (مايو 2004). "The Ensembl automatic gene annotation system". Genome Research. ج. 14 ع. 5: 942–950. DOI:10.1101/gr.1858004. ISSN:1088-9051. PMC:479124. PMID:15123590.
- ↑ Ruffier، Magali؛ Kähäri، Andreas؛ Komorowska، Monika؛ Keenan، Stephen؛ Laird، Matthew؛ Longden، Ian؛ Proctor، Glenn؛ Searle، Steve؛ Staines، Daniel (يناير 2017). "Ensembl core software resources: storage and programmatic access for DNA sequence and genome annotation". Database. ج. 2017 ع. 1: bax020. DOI:10.1093/database/bax020. PMC:5467575. PMID:28365736.
- ↑ Hubbard، T. J. P.؛ Aken، B. L.؛ Ayling، S.؛ Ballester، B.؛ Beal، K.؛ Bragin، E.؛ Brent، S.؛ Chen، Y.؛ Clapham، P. (يناير 2009). "Ensembl 2009". Nucleic Acids Research. ج. 37 ع. Database issue: D690–697. DOI:10.1093/nar/gkn828. ISSN:1362-4962. PMC:2686571. PMID:19033362.
- ↑ Howe، Kevin L.؛ Contreras-Moreira، Bruno؛ De Silva، Nishadi؛ Maslen، Gareth؛ Akanni، Wasiu؛ Allen، James؛ Alvarez-Jarreta، Jorge؛ Barba، Matthieu؛ Bolser، Dan M. (8 يناير 2020). "Ensembl Genomes 2020-enabling non-vertebrate genomic research". Nucleic Acids Research. ج. 48 ع. D1: D689–D695. DOI:10.1093/nar/gkz890. ISSN:1362-4962. PMC:6943047. PMID:31598706.
- ↑ "The Ensembl Core Software Libraries". Genome Research. ج. 14 ع. 5: 929–933. فبراير 2004. DOI:10.1101/gr.1857204. PMC:479122. PMID:15123588.
- ↑ "Species List". uswest.ensembl.org. مؤرشف من الأصل في 2022-08-06. اطلع عليه بتاريخ 2022-08-05.